米兰-BPO行业整合方案提供者
专业化、科技化、国际化;高标准、广覆盖、全流程
了解更多
2024年10月23日,北京年夜学计较机学院张成与定量生物学中间钱珑结合研究团队与合作者,在国际学术期刊Nature上颁发题为“Parallel molecular data storage by printing epigenetic bits on DNA” 的研究论文,初次提出了一种基在并行写入策略的DNA存储方式。该手艺不依靠在主流的“从头合成”写入线路道理,经由过程DNA自组装与选择性酶促甲基化的组合道理,可将“表不雅比特”(epi-bit,5-甲基胞嘧啶)编码的数字信息并行打印在DNA份子上。同时,研究中还功实现了小我订制DNA存储示例,证实了便捷的散布式DNA存储利用潜力。该方式的成立,不但为实现了快速、低本钱的年夜范围份子数据存储奠基了手艺根本,还为将来DNA存储的成长供给了全新思绪。
 论文截图
论文截图 ?
年夜数据时期,全球数据大水对数据存储手艺提出了严重挑战。DNA份子具有超高的数据存储密度和超长命命,已成为备受注视的倾覆性存储介质。但是,传统DNA存储依靠“从头合成”的信息写入线路,在本钱和速度上面对庞大挑战。分歧在传统手艺线路,张成、钱珑结合团队开辟的“表不雅比特(epi-bit)”DNA存储操纵预制的DNA模板和份子活字块,经由过程DNA自组装介导的份子信息排版,经选择性酶促甲基润色转移,实现了份子级“活字印刷”信息打印。
 图1. 表不雅比特DNA存储信息暗示方式和Nature相干专题报导
图1. 表不雅比特DNA存储信息暗示方式和Nature相干专题报导 ?
团队在尝试中,将中国汉朝“白虎”瓦当和国宝年夜熊猫“飞云”的高清图片成功写入DNA份子中,数据量跨越27.5万比特,比拟此前颁发的其他非传统DNA存储手艺,数据范围晋升跨越300倍。信息读取利用便携式纳米孔测序仪,实现了对DNA模板上复杂表不雅比特信息的高通量读取,并经由过程单次超240种分歧润色模式的并行解析,无损还原了原始数据。尝试成果验证了该立异型份子存储手艺的可行性和正确性,还展现了表不雅比特的不变性。
值得存眷的是,团队还展现了这项手艺的散布式存储利用潜力。在小我定制DNA存储尝试中,约请了北京年夜学、华北电力年夜学等单元60名布景普遍的青年自愿者,由他们在平常情况下,将私家数据亲手写入DNA并由小我保留,相干数据直到利用时才被读取,可有用保障小我数据的隐私与平安。这类散布式DNA存储体例,不但能极年夜下降DNA存储的利用门坎,且保障了数据隐私,有望鞭策DNA存储的小我利用。
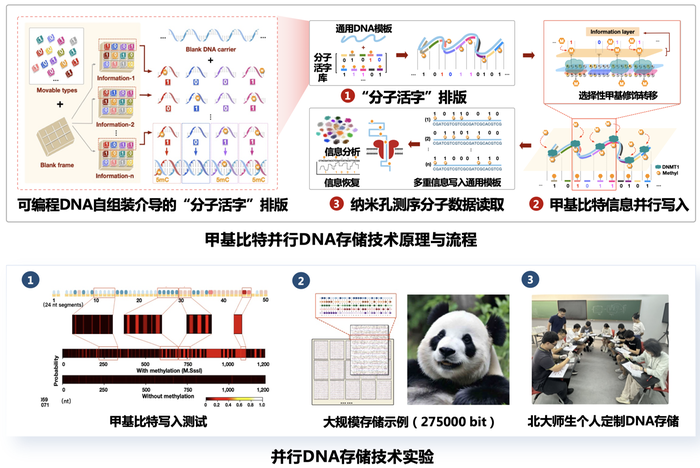 图2. 表不雅比特DNA存储道理流程和尝试成果
图2. 表不雅比特DNA存储道理流程和尝试成果 ?
表不雅比特DNA存储框架为年夜范围数据存储供给了全新的解决方案,有望冲破DNA存储的本钱和速度壁垒。该手艺的开辟,还揭示了非传统份子比特在数据存储中的怪异优势,为将来新型份子信息处置系统的研发奠基了根本。张成、钱珑、北京年夜学传授欧阳颀和美国亚利桑那州立年夜学传授颜颢为本文的配合通信作者。吴燃峰、孙法家和林艺生为本文共一作者。北京年夜学计较机学院和定量生物中间为本文第一通信单元。本研究取得了德国斯图加特年夜学刘娜团队、成都瀚辰光翼科技有限责任公司、年夜连理工年夜学张强团队、华北电力年夜学杨静团队的鼎力撑持。本研究的iDNAdrive尝试取得了北京年夜学2024年iGEM团队的鼎力撑持。该工作获得了国度重点研发打算、国度天然科学基金、军委设备研究项目、北京年夜学-鲲鹏昇腾科教立异出色中间项目标帮助。
Nature杂志同期news views栏目专题报导(https://www.nature.com/articles/d41586-024-03312-6 )。
(原题目:张成、钱珑结合研究团队与合作者在《天然》发文,初次提出基在并行写入策略的DNA存储方式)
特殊声明:本文转载仅仅是出在传布信息的需要,其实不意味着代表本网站不雅点或证实其内容的真实性;如其他媒体、网站或小我从本网站转载利用,须保存本网站注明的“来历”,并自大版权等法令责任;作者假如不但愿被转载或联系转载稿费等事宜,请与我们联系。